1 Documentos científicos
En su actividad diaria, los estudiantes, académicos y especialistas científicos producen gran cantidad de documentación de todo tipo: notas de laboratorio, apuntes, memorandos, informes técnicos y, sobre todo, artículos científicos para publicar sus descubrimientos y avances en un área de conocimiento. Normalmente, la creación de este tipo de documentos científicos conlleva una gran cantidad de tareas que involucran diferentes herramientas y posibles puntos de fallos.
La Figura 1.1 muestra una descripción general esquemática de un proceso de trabajo clásico para la creación de documentos científicos. Con frecuencia, el elemento principal es un archivo maestro de procesador de textos (Word, OpenOffice/LibreOffice, etc.), una página web o un fichero LaTeX (si vamos a crear un documento PDF) que recoge todo el contenido.
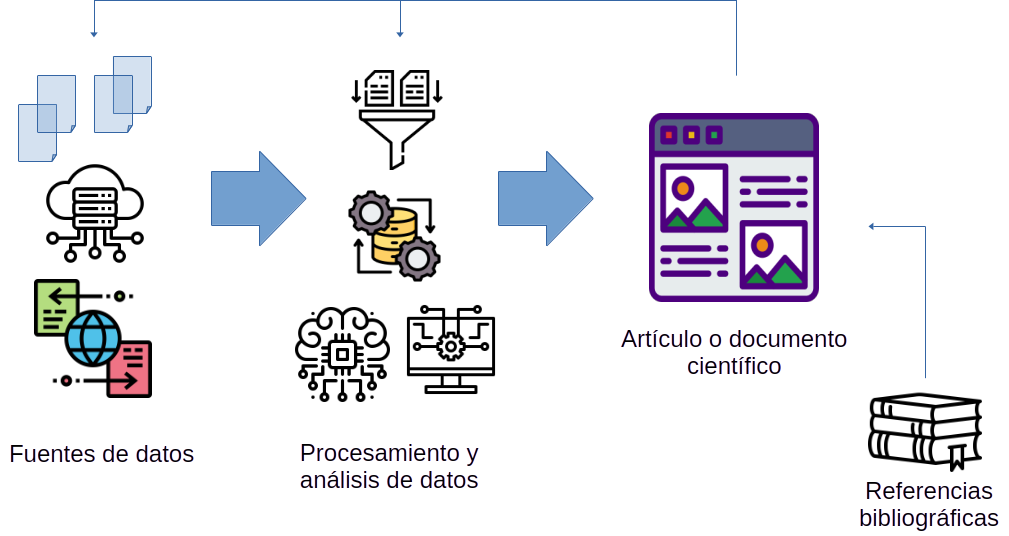
Este archivo maestro se va llenando de contenido que procede de diversas fuentes, como por ejemplo:
- figuras y esquemas generados manualmente o mediante código software (como gráficos de visualización de datos);
- tablas y resúmenes que describen conjuntos de datos y resultados;
- resultados y evaluación del rendimiento de modelos o algoritmos; estadísticos o de aprendizaje automático;
- fórmulas y ecuaciones matemáticas;
- tablas de datos y otra información de utilidad;
- referencias bibliográficas (normalmente generadas con ayuda de algún programa de gestión de información bibliográfica).
Muchos de estos elementos fuerzan a los usuarios a ejecutar una y otra vez herramientas y programas externos, procedimientos y otras tareas para incorporar luego los nuevos resultados al fichero maestro. Debemos admitir que este proceso, en su mayor parte manual, además de tedioso puede ser muy propenso a que cometamos errores o descuidos. “¡Espera! He olvidado actualizar la Figura 1”. “¿Seguro que estos son los últimos resultados de evaluación del modelo M?” “¿Has comprobado que hemos cargado la última versión del fichero de datos D?” Estas son preguntas comunes que surgen en el día a día de los equipos científicos.
Sin embargo, sería genial si no fuese necesario realizar todo ese proceso manual y, en ocasiones, muy frustrante de forma manual. ¿Tenemos alguna alternativa para evitarlo? Sí, la tenemos. La respuesta a nuestras necesidades nos la brinda un concepto muy poderoso: la programación literaria.
1.1 Programación literaria
El concepto de programación literaria fue acuñado por el profesor Donald E. Knuth (1984). Sí, no has leído mal, hace más de 40 años. Este concepto establece que debería ser posible integrar, en un solo documento científico, texto formateado y resultados de la ejecución de código software para componer dicho documento de forma dinámica. Entonces, ¿por qué hemos tardado tanto en poner en práctica esta idea? La visión de Knuth, aunque muy adelantada a su tiempo, era correcta, pero la tecnología de la época no permitía ponerla en práctica.
Sin embargo, hoy día contamos con todos los elementos indispensables para llevar esta idea a la práctica. Es más, contamos con una herramienta, Quarto, que nos va a permitir automatizar y gestionar todo el proceso de creación de documentos de programación literaria de forma rápida y fiable.
1.2 Investigación reproducible
Durante muchas décadas, el método científico se ha basado en la publicación de investigaciones que describen el resultado de análisis de datos y experimentos. En todos los casos, resulta fundamental poder confiar en las condiciones, los datos recabados, el método de análisis y de ejecución de los experimentos así como en las herramientas de diversas clase, incluído el software, que los autores de la publicación han empleado para llevarla a cabo.
Sin embargo, los numerosos avances experimentados en los últimos años en las herramientas y métodos de análisis permiten que ahora sea mucho más sencillo comprobar el resultado de estos análisis. Podríamos suponer que esto facilita mucho el trabajo de científicos y científicas, pero en realidad sucede todo lo contrario. Veamos algunos ejemplos:
Oncología (Begley & Ellis, 2012): El Departamento de Biotecnología de la firma Amgen (Thousand Oaks, CA, EE.UU.) sólo pudo confirmar 6 de un total de 53 artículos de investigación emblemáticos publicados en este área. Por su parte, Bayer HealthCare (Alemania) tan sólo pudo validar un 25% de los estudios analizados.
Psicología (Wicherts et al., 2006): El 73% de los autores de un total de 249 artículos publicados por la APA no respondieron en un periodo de 6 meses a las preguntas y requerimientos formulados acerca de los datos que emplearon en sus investigaciones.
Economía y finanzas (Burman et al., 2010): La comparación de diferentes paquetes software aplicados en la ejecución de varios análisis de modelos financieros y estadísticos refleja que cada uno de dichos paquetes produce resultados muy distintos empleando las mismas técnicas estadísticas directamente aplicadas sobre datos idénticos a los empleados en la publicación original.
De hecho, han llegado a aparecer artículos que sugieren que buena parte de los resultados publicados en áreas como Medicina podrían no ser del todo fiables (Ioannidis, 2005). Como resultado de todos estos hallazgos recientes, se ha generado en toda la comunidad científica e investigadora una gran polémica, acompañada de una profunda crisis de confianza.
A pesar de todo, como muy bien recoge una conocida tira cómica sobre el mundo académico y la investigación (ver Figura 1.2), el proceso de desarrollo de las publicaciones científicas se basa primordialmente en la revisión continua de los métodos y resultados (empezando por los propios estudiantes y sus supervisores).

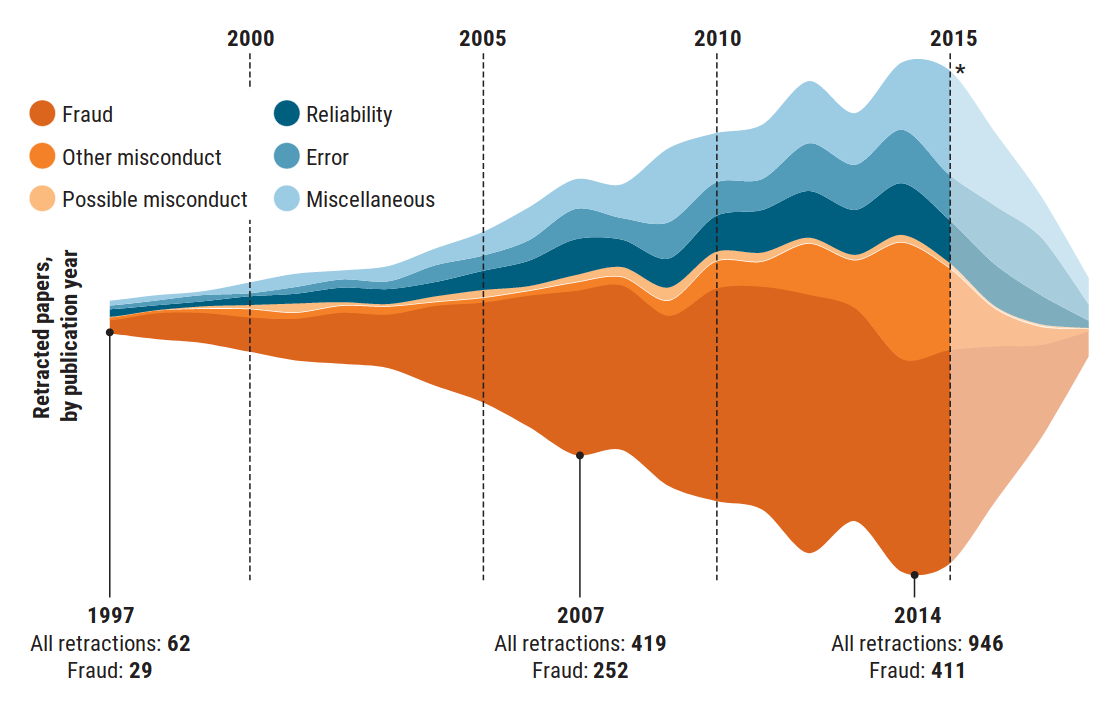
La Figura 1.3 muestra un gráfico publicado en la prestigiosa revista Science Magazine (Brainard et al., 2018), que representa los datos sobre evolución del número de artículos de investigación retractados o retirados por diveresas causas, entre 1997 y 2014. En este gráfico, podemos constatar cómo la mejora de las herramientas y la mayor disponibilidad de recursos permite analizar y revisar un mayor volumen de publicaciones y análisis, lo que permite detectar un mayor número de casos problemáticos.
1.2.1 Reproducibilidad y replicabilidad
Se habla con frecuencia de reproducir y replicar un análisis de datos o un experimento científico (Leek & Peng, 2015). Sin embargo, se pueden citar numerosas evidencias que demuestran que existen definiciones incompatibles sobre estos dos términos y otros relacionados con ellos (Barba, 2018). Mucho cuidado, por tanto, porque dependiendo de la comunidad científica o el campo de conocimineto en que nos encontremos, el significado de estos dos terminos puede llegar a ser incluso enteramente opuesto a su definición aceptada en otras áreas 1. Aquí vamos a ceñirnos a la definición aceptada en un gran número de áreas, incluyendo estadística o computación científica (véase Barba, 2018, p. 33):
Reproducibilidad: Se define como la capacidad para recomputar los resultados de un análisis, con los mismos datos que se emplearon en el análisis original, y conociendo los detalles de la secuencia (workflow o pipeline) de operaciones de componen dicho análisis. Se debe poder garantizar ciertas premisas:
Si usamos las mismas herramientas (e.g. R, un cierto listado de paquetes, las mismas versiones de todos los paquetes y dependencias), así como el mismo código (scripts de R) sobre los mismos datos, los resultados y conclusiones han de ser consistentes con los del análisis original.
Los autores del análisis original deben proporcionar todos los elementos (datos, código y procedimiento empleado) para permitir que el análisis sea reproducible (Barba, 2018).
Replicabilidad: Se define como la capacidad para realizar un experimento o análisis independiente del original, que aborde el mismo objetivo pero sobre un conjunto de datos diferente del empleado en el estudio inicial. En caso de que los resultados no sean consistentes, será necesario realizar más réplicas y armonizar los resultados y conclusiones por medio de técnicas adecuadas, como por ejemplo el meta-análisis.
1.2.2 Niveles de replicación
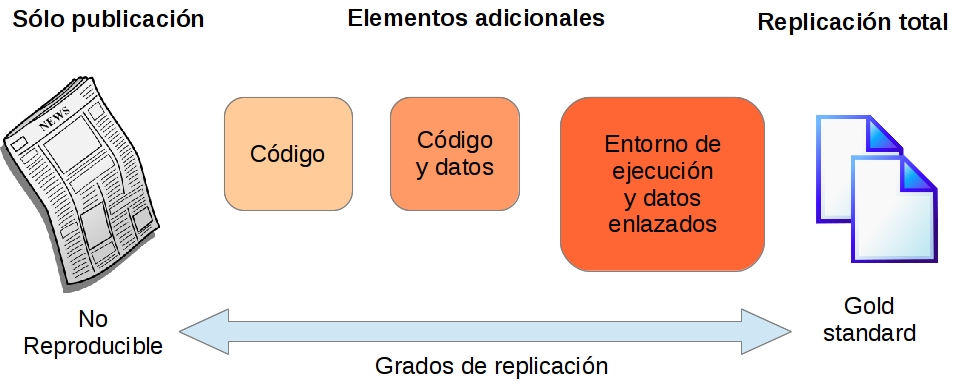
En función de los elementos publicados por los autores del estudio original, así como del grado de detalle con el que se describe el proceso para llevar a cabo el estudio, los pasos que se han seguido y las herramientas empleadas, tenemos diferentes niveles de replicabilidad o reproducibilidad, representados en la Figura 1.4.
No reproducible: No se proporcionan datos, código ni ninguna descripción concreta sobre la implementación del estudio o análisis. Muchas publicaciones científicas ya no aceptan publicar artículos en estas condiciones.
Código o Datos: Un buen número de editoriales solicitan que los conjuntos de datos empleados en el análisis o estudio de la publicación sean accesibles mediante una URL, bien porque estén disponibles en un repositorio público o bien porque los autores del artículo lo han publicado. Así mismo, muchas publicaciones exigen que el código software para llevar a cabo el análisis también esté accesible públicamente, en un repositorio de código abierto o bien en un proyecto de un servicio de control de versiones con acceso libre.
Código y datos: Lo ideal es que tanto el código como los datos estén públicamente accesibles a disposición de quien los quiera examinar o bien utilizar para reproducir los resultados (validación) o replicar el análisis con otros datos u otros casos.
Entorno de ejecución y datos enlazados: Un paso más para facilitar la reproducibilidad de los estudios consiste en publicar archivos de código y metadatos con información más precisa sobre el lenguaje de programación, los paquetes software empleados y cualquier otra dependencia necesaria para llevar a cabo el mímo estudio o análisis. Otra variante para facilitar la reproducibilidad es la de encapsular el código y las dependencias en un contenedor virtual ya preconfigurado, que se pueda descargar y ejecutar directamente.
Gold standard: El nivel más avanzado consistiría en documentar todos los procedimientos realizados durante el estudio o análisis, incluyendo la codificación de las tareas de obtención, limpieza y preparación de los datos, así como la generación de gráficas de visualización de resultados o cualquier otro resultado derivado del estudio.
1.2.3 Herramientas para la replicabilidad
Ciertas tecnologías y herramientas que se han refinado y sofisticado durante los últimos años están permitiendo facilitar la replicabilidad del procesamiento y análisis de datos.
- Sistemas de Control de Versiones para código software (SCV): herramientas como Git, Mercurial y servicios web como GitHub o GitLab han popularizado la creación y publicación de proyectos que permiten gestionar el código software que se ha creado, controlando los cambios y las versiones liberadas. Los servicios web integran, además, un buen número de herramientas para dar soporte a diferentes facetas del proceso de desarrollo de software, tales como generación de documentación, manuales y ejemplos, informes de error y peticiones de mejoras, integración continua y despliegue continuo (CI/CD), testeo sistemático del código generado, etc. Si todavía no te has planteado en qué puede beneficiarte utilizar una herramienta de control de versiones de código fuente, echa un vistazo a la Figura 1.5 en la que revivirás una situación lamentablemente muy habitual entre los investigadores y científicos que desarrollan soluciones software.
Virtualización de software y contenedores: en un entorno tecnológico dominado por la contratación y despliegue de infraestructura de computación y servicios en arquitecturas en la nube (cloud computing), las herramientas de empaquetado y virtualización de aplicaciones y servicios software que pueden instalarse y desplegarse en poco tiempo han revolucionado la forma en la que se publican y gestionan los productos software, incluidos los de procesamiento y análisis de datos.
Control de versiones de datos: De forma análoga a los SCV para código fuente, está apareciendo software para aplicar los mismos principios a los ficheros de datos. De esta forma, podemos controlar diferentes versiones de cada archivo de datos, modificaciones efectuadas en los mismos, etc. Una de estas herramientas es Data Version Control (DVC), que permite versionado de datos y modelos. Como resultado, podemos saber en todo momento qué versión de los datos y qué listado de features se han incluido en cada modelo considerado durante el análisis, manteniendo integrada la información descriptiva sobre estos tres componentes esenciales que siempre deben ir cohesionados.
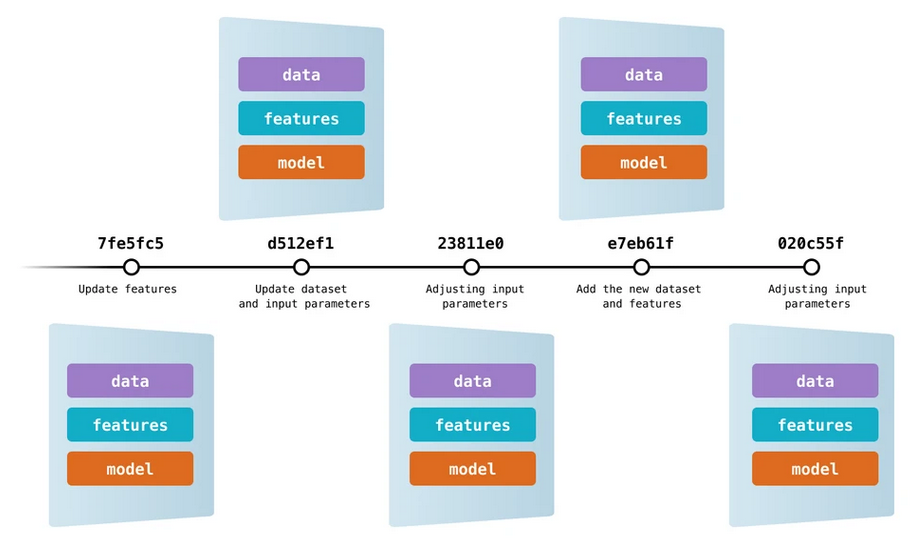
Gestión de modelos y experimentos: Otro tipo de herramienta de gestión de proyectos de aprendizaje automático es la que permite la organización, seguimiento, comparativa y selección de los experimentos y modelos que hemos realizado. Uno de los ejemplos destacados más recientes es ML Flow, que proporciona soporte para ajuste, evaluación y optimización de modelos, despliegue de los mismos en entornos en producción, creación de un registro de modelos pre-entrenados, etc. Por supuesto, es posible combinar este tipo de herramientas con otras como DVC, creando como resultado un entorno integral de gestión de nuestros proyectos.
Creación y gestión de pipelines de procesamiento de datos: el último elemento indispensable en todo proyecto de procesamiento y análisis de datos que deba cuidar la escalabilidad es una herramienta para creación y gestión de flujos de procesamiento y análisis o pipelines de datos. El conjunto de todos los pipelines de nuestro proyecto componen el workflow general del mismo. A estas herramientas se las conoce como orquestadores de datos o de flujos de trabajo. En esta categoría, contamos tanto con herramientas muy potentes y llenas de características como Apache Airflow o Prefect como con otras más sencillas y directas como Luigi.
Por supuesto, la comunidad de R no se ha mantenido ajena a estas nuevas tendencias, muy en particular la iniciativa R OpenSci, dentro de la cual encontramos muchos paquetes (publicados en el repositorio oficial CRAN) que cubren diversos aspectos del trabajo científico, incluyendo la gestión de pipelines y workflows mediante el paquete targets.
1.3 Quarto para publicaciones científicas
Ahora que ya conocemos el concepto fundamental sobre el que se asienta el funcionamiento de Quarto y su aplicación para conseguir un mayor nivel de reproducibilidad y transparencia en nuestro proceso científico, vamos a explicar con más detalle el proceso que sigue Quarto para componer un documento. La Figura 1.7 presenta un esquema con el proceso de creación del documento y los elementos y herramientas que entran en juego para conseguirlo.
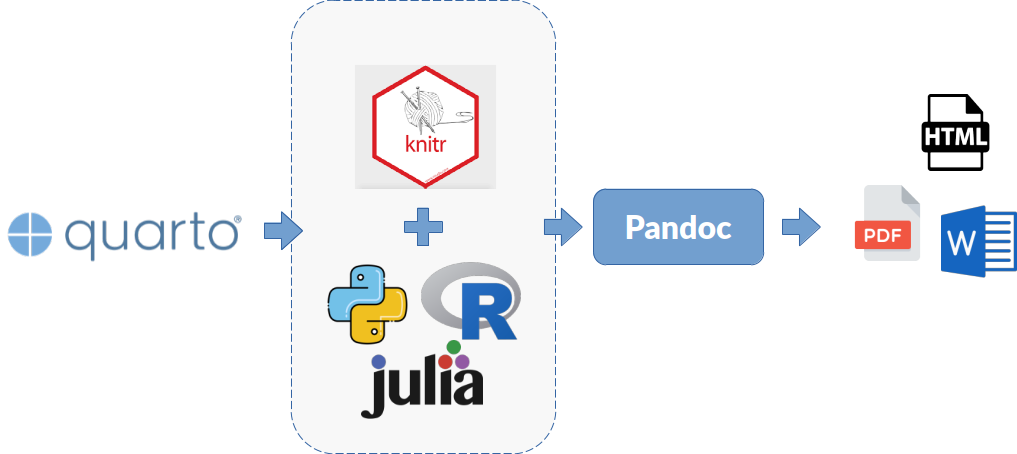
- Quarto: un software que permite crear documentación científica siguiendo los principios de la programación literaria.
- Knitr y lenguaje de programación: el paquete
knitrse encarga de la conexión con un intérprete de un lenguaje de programación (R, Python, Julia) que se pueda ejecutar en un entorno REPL, para poder ejecutar fragmentos de código software integrados en el documento y generar como resultado contenido en formato Markdown. - Markdown (contenido formateado): lenguaje de marcado de contenido textual que permite formatear de forma sencilla la información de nuestros documentos creados con Quarto.
- Pandoc (traductor universal de formatos documentales): este software recibe el contenido ya formateado usando el estándar Markdown, para convertirlo en el tipo de salida seleccionado. Existen varias opciones disponibles: HTML, PDF o Word, así como diapositivas, websites o paneles interactivos (dashboards).
1.4 Instalación de Quarto
Para instalar la última versión del software Quarto en nuestro sistema, dirigimos nuestro navegador web a la página https://quarto.org/docs/get-started/. Aquí, descargamos e instalamos el archivo correspondiente a nuestro sistema operativo.
En este momento, la última versión de Quarto disponible es la 1.5.57.
Por defecto, el formato de salida de los documentos generados con Quarto es HTML. Si queremos generar documentos en PDF, necesitamos tener instalada una distribución LaTeX. Para más información, consulta la Sección 3.3.
Entre los ejemplos más importanes de definiciones que contradicen las que damos en este taller, están las adoptadas por la Federation of American Societies for Experimental Biology (FASEB), en inmunología y microbiología, así como las adoptadas por la Association for Computer Machinery (ACM) en ciencias de la computación.↩︎